DEG解析で'row.names'に重複した名前を付けられない場合の解決法
先日、読者の方から「Differential Expression Analysisでマトリックスをインポートする際に行名の重複を警告される」というコメントを拝見しましたので、その理由と最もシンプルな解決策をご説明します。
その理由は プローブと遺伝子は多対一の関係にあり、例えば A と B は共に遺伝子 AB を指している場合があります。通常の遺伝子マイクロアレイの発現行列では、プローブで表現される発現行列は行名の重複がない。これは、Rのルールでは行名を一意な識別子とみなしており、行名を使ってデータを取得する際に、2つの行が同じ名前であれば、行名重複の必要性をRが認識しないためである。Rは、行名を使用してデータを取得する際に、どの行が必要なのかわからなくなるのです。
解決策 通常、異なるプローブから得られた遺伝子記号を一定のルールに従って1つの行にまとめればよい。平均値、中央値、最大値、最小値など、ニーズに応じて作ればよい。この操作はRで行うことができるが、多少のプログラミングスキルが必要である。ここでは、Excelを使って、同じ行を平均値にマージする例を示します。
1. これは、行名が繰り返される式行列で、サンプルは1つだけです。

2. 新しいシートを作成し、左上にあるピボットテーブルをクリックします。
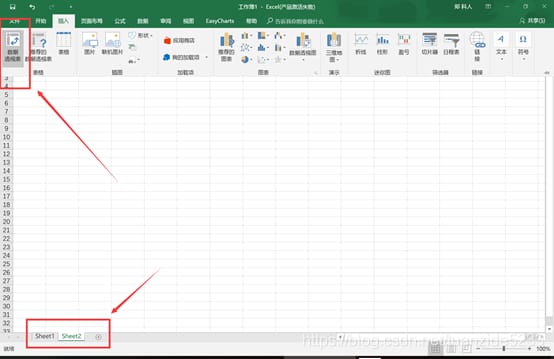
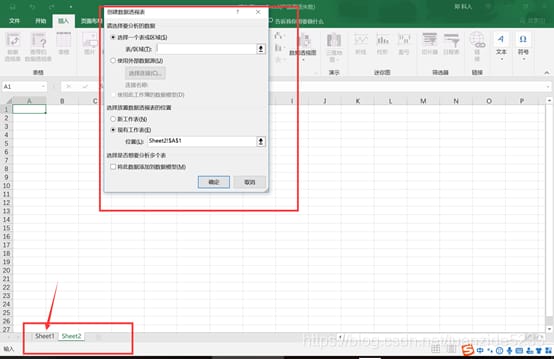
3. ダイアログボックスが表示されたら、sheet1 に戻り、範囲を選択します。
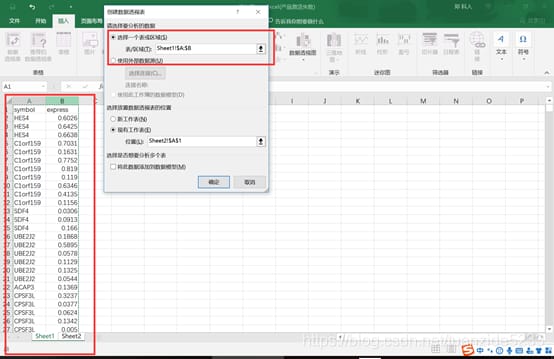
4. sheet2 の必要な列をチェックします。
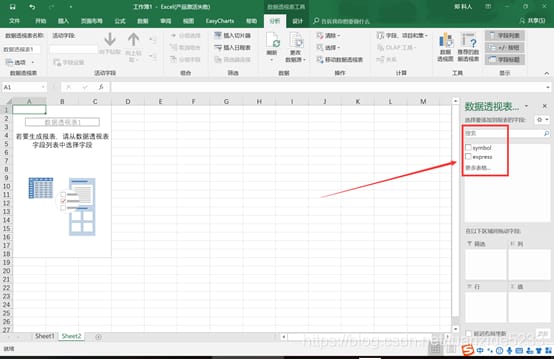
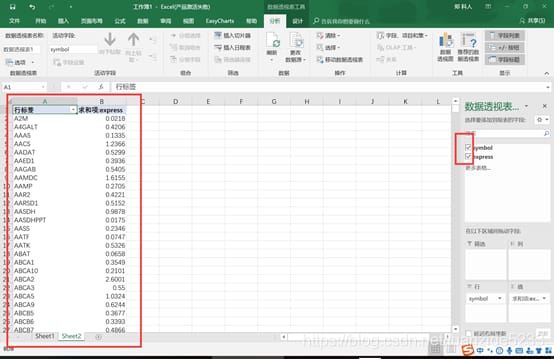
5. 5. 合計項目(B1)をダブルクリックし、新しいダイアログボックスで平均を選択すると、同じ名前の行が平均に 従って各サンプルについて計算されます。
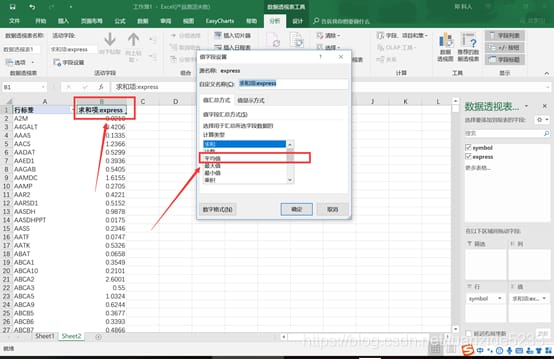
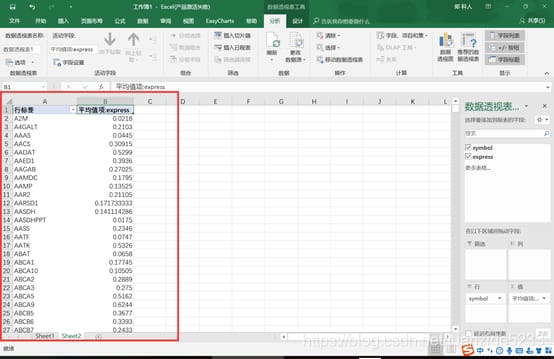
6. このアプローチでは、一部の遺伝子記号が日付指定されてしまう可能性があることに注意が必要です。しかし、一般的には、プログラミングの弱い実務者にとっては、まだ使える、信頼できる方法である。
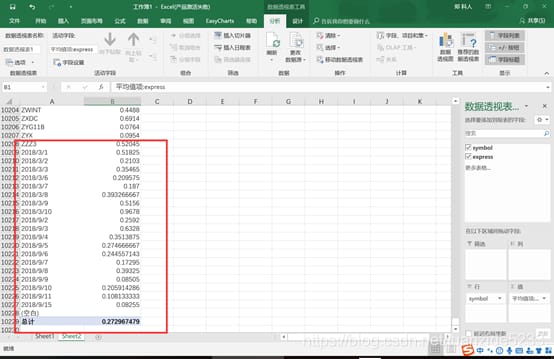
7. 変換した行列をRにインポートすれば問題ない。
GEOチップデータの差分発現解析にlog2処理が必要な理由
https://blog.csdn.net/tuanzide5233/article/details/88542805
GEOマイクロアレイデータの差分発現解析にlog2が必要かどうか、正規化の問題点
https://blog.csdn.net/tuanzide5233/article/details/88542558
差分表現マトリックス作成チュートリアル
https://blog.csdn.net/tuanzide5233/article/details/83659768
微分表現のヒートマッププロットは、以下のページで詳しく説明しています。
https://blog.csdn.net/tuanzide5233/article/details/83659501
edgeRを用いたRNAseqデータの差分発現解析に関するチュートリアル
https://blog.csdn.net/tuanzide5233/article/details/88785486
微分発現解析(DEG)時に'row.names'に重複した名前を表示させないための解決策です。
https://blog.csdn.net/tuanzide5233/article/details/86568155
生存時間解析チュートリアルシリーズ (I) Bioshiners Toolboxを用いた生存時間解析
https://blog.csdn.net/tuanzide5233/article/details/83685403
エンリッチメント解析とタンパク質相互作用ネットワーク(PPI)の可視化 シストケープ入門ガイド
https://blog.csdn.net/tuanzide5233/article/details/88048439
Vennプロットの上級編:Upsetプロットの入門編ハンズオンコード詳細 - UpSetRパッケージの紹介
https://blog.csdn.net/tuanzide5233/article/details/83109527
R言語ggplot2パッケージを用いたパスウェイエンリッチメント解析バブルプロット(Bubble plots)のプロット:データ構造とコード
関連
-
RStudio の "plot.new() : figure margins too large" 問題を解決する。
-
二項演算子への非数値引数を報告するR言語エラー
-
R: エラー: $ 演算子は原子ベクトルに対して無効です。
-
R LanguageError in hist.default() : 'x' は数値でなければなりません.
-
R言語エラー:図の余白が大きすぎる 解決方法
-
R plot.new() のエラー : 図形の余白が大きすぎる
-
[解決策】 plot.new() のエラー:図の余白が大きすぎる。
-
ggplot2 からグリッドと背景色を削除する。
-
Rでエラー:単項演算子への引数が無効
-
R - ユークリッド距離の計算を簡単にする方法
最新
-
nginxです。[emerg] 0.0.0.0:80 への bind() に失敗しました (98: アドレスは既に使用中です)
-
htmlページでギリシャ文字を使うには
-
ピュアhtml+cssでの要素読み込み効果
-
純粋なhtml + cssで五輪を実現するサンプルコード
-
ナビゲーションバー・ドロップダウンメニューのHTML+CSSサンプルコード
-
タイピング効果を実現するピュアhtml+css
-
htmlの選択ボックスのプレースホルダー作成に関する質問
-
html css3 伸縮しない 画像表示効果
-
トップナビゲーションバーメニュー作成用HTML+CSS
-
html+css 実装 サイバーパンク風ボタン
おすすめ
-
R 描画エラー plot.new() : 図形の余白が大きすぎる
-
R言語です。「接続を開くことができません」解決策
-
R言語のエラーメッセージと関連する解決策
-
R: hclust(d, method = method)でのエラー : 外部関数呼び出しは NA/NaN/Inf(arg10) を持つことができません。
-
R: 環境と変数のスコープ問題
-
R - よくあるエラーとその原因 - 注意事項
-
SocketTimeoutExceptionです。読み込みがタイムアウトしました
-
R read.table Error:埋め込まれたヌルが含まれているようです。
-
[R] is.data.frame(x) のエラー : (リスト) オブジェクトを 'double' 型に強制できない。
-
Rの警告 "条件の長さが1より大きいので、最初の要素しか使えない "に対する解決策